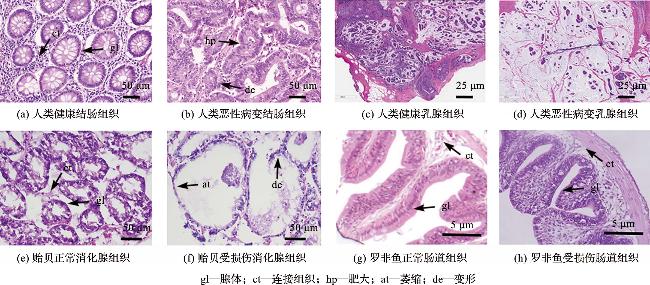
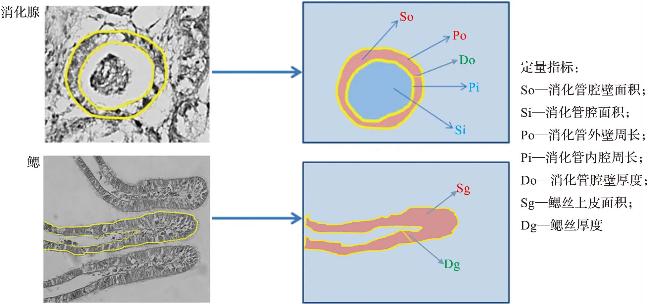
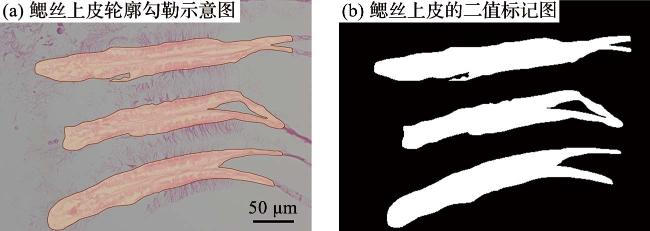
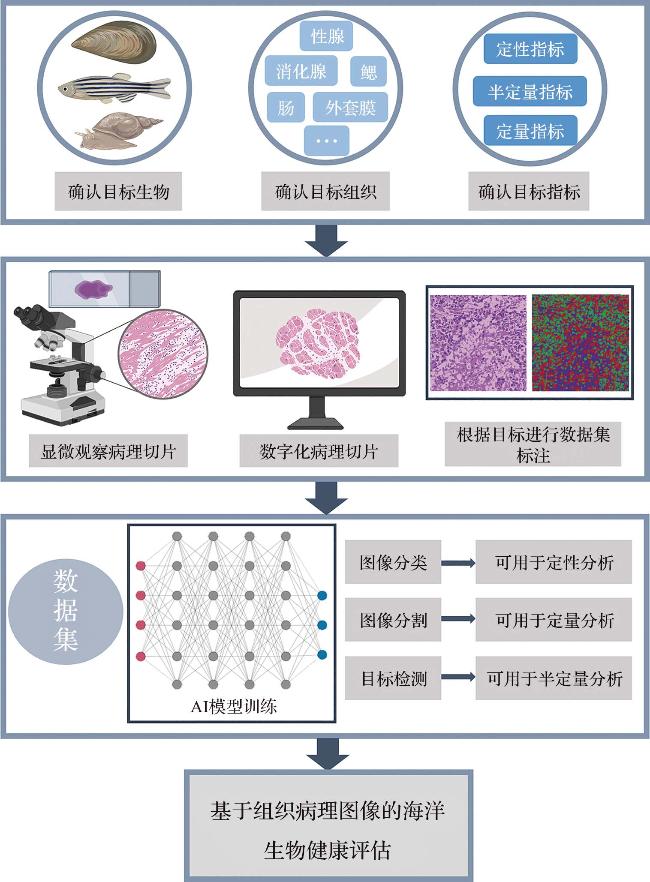
上述基于贻贝组织病理特征的生物健康评价多停留在定性层面,无法建立环境变化与生物健康损伤之间的剂量-效应关系;并且,由于组织病理特征识别主要依赖研究人员通过目视进行图像判读,存在主观性强、错误率高的缺陷
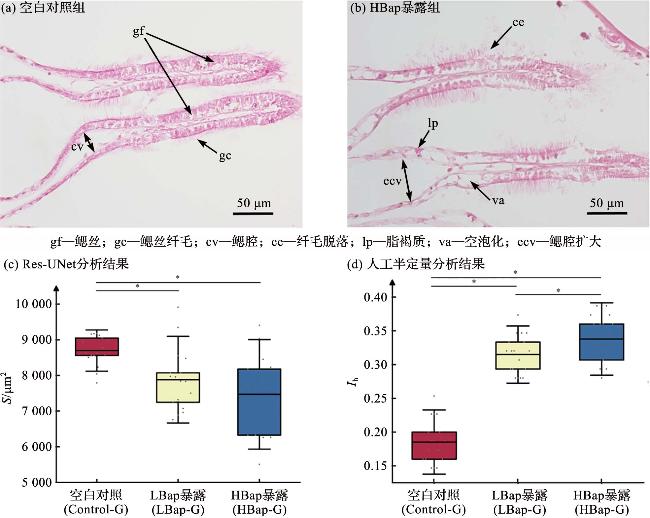
[32]。针对贻贝中常见的组织损伤类型,研究人员开发了表征生物健康水平的半定量指标(
Ih):首先对组织病理图像进行盲读,统计出每张图像中出现的病理损伤种类及出现频次,接着对不同病理特征所代表的损伤程度进行赋值(如血细胞浸润赋值1,纤维化赋值2,坏死赋值3等),再通过加权计算获得每张组织病理图像的
Ih值
[33]。CUEVAS等
[34]采用贻贝(
M.galloprovincialis)消化腺和性腺的半定量指标表征的健康状态来评估西班牙巴斯克海岸的污染水平,结果显示生活在污染最严重区域的贻贝,两种组织中的
Ih值均显著高于生活在其他区域的贻贝,并且消化腺始终比性腺受到的损害更严重。COPPOLA等
[35]在实验室进行聚乙烯亚胺官能化-氧化石墨烯(graphene oxide functionalized with polyethyleneimine,GO-PEI)和汞两种污染物的单独或复合暴露,发现贻贝(
M. galloprovincialis)鳃和消化腺中的
Ih指标均明显升高,表明当环境中存在这两种污染物时,会对贻贝健康造成损害。尽管
Ih值可以通过定量数据反映环境变化对贻贝健康的损害程度,但是半定量指标本质上也是基于对病理图像的主观判断
[36]。