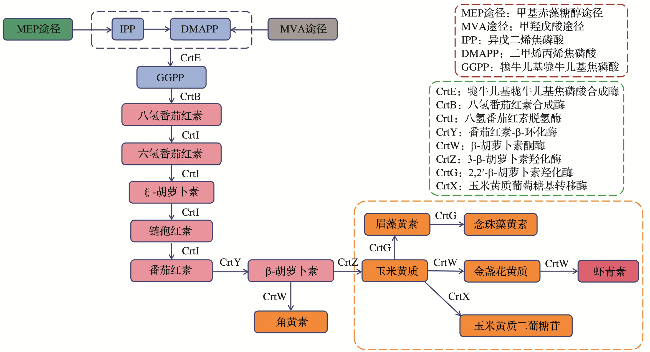
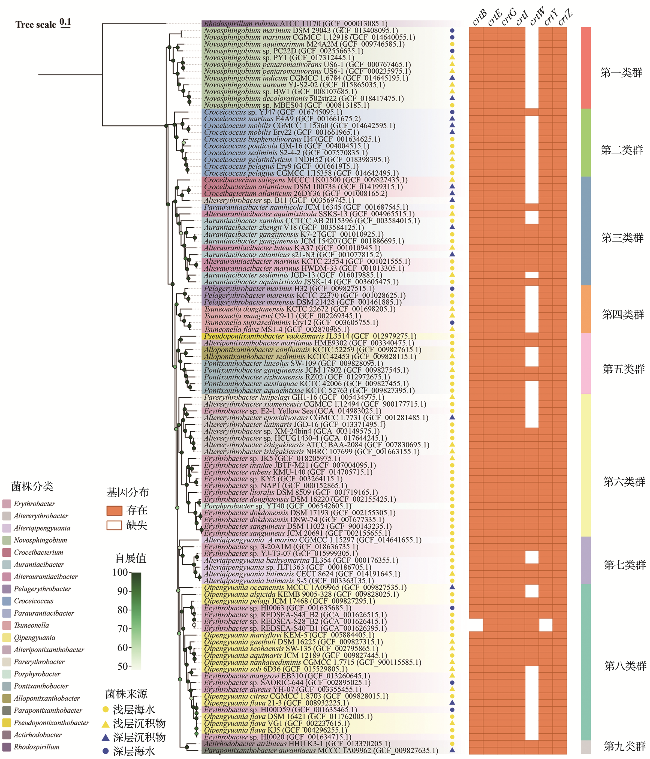
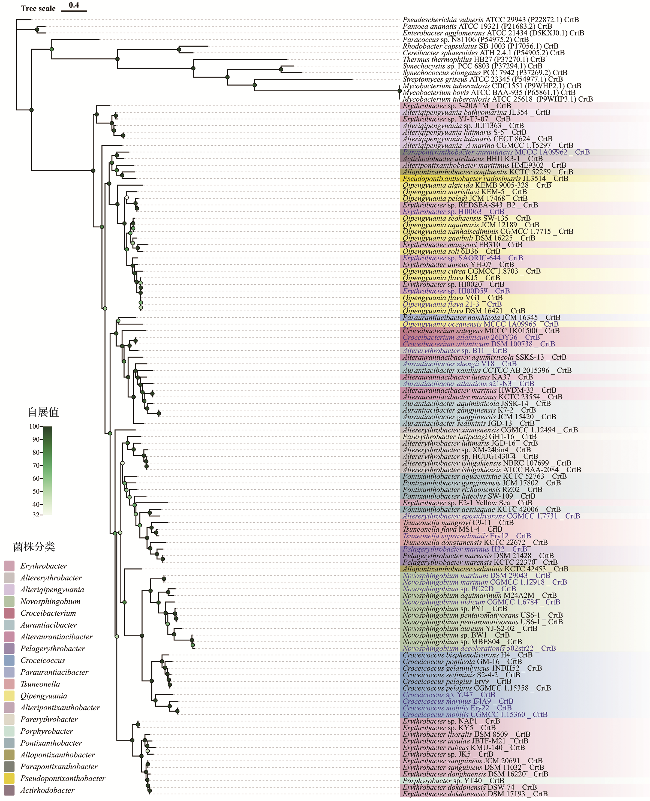
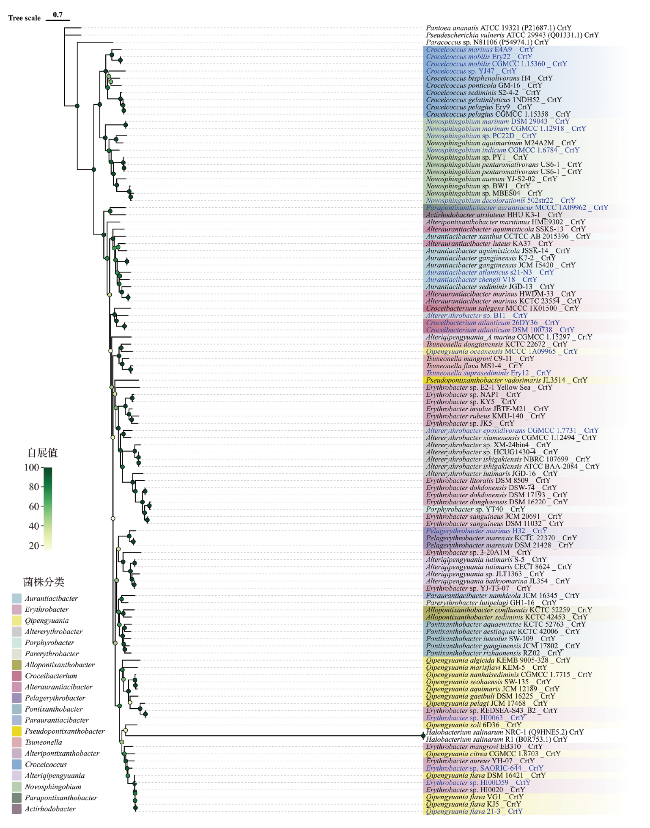
| 1 | GCA_001626395.1 | Erythrobacter sp. REDSEA-S40_B1 | 2 688 120 | 63.7 | 7 | 浅海海水 |
| 2 | GCA_001626415.1 | Erythrobacter sp. REDSEA-S28_B2 | 2 687 817 | 63.7 | 8 | 浅海海水 |
| 3 | GCA_001626515.1 | Erythrobacter sp. REDSEA-S43_B2 | 2 871 770 | 63.6 | 18 | 浅海海水 |
| 4 | GCA_003149575.1 | Altererythrobacter sp. XM-24bin4 | 2 534 571 | 56.8 | 12 | 浅海海水 |
| 5 | GCA_014983025.1 | Erythrobacter sp. E2-1 Yellow Sea | 2 518 489 | 57.4 | 31 | 浅海海水 |
| 6 | GCA_017644245.1 | Altererythrobacter sp. HCUG1430-4 | 2 509 776 | 56.8 | 9 | 浅海海水 |
| 7 | GCF_000152865.1 | Erythrobacter sp. NAP1 | 3 266 173 | 61.0 | 2 | 浅海海水 |
| 8 | GCF_000176355.1 | Alteriqipengyuania bathyomarina JL354 | 3 273 334 | 65.0 | 68 | 浅海海水 |
| 9 | GCF_000186705.1 | Alteriqipengyuania sp. JLT1363 | 3 117 324 | 64.9 | 26 | 浅海海水 |
| 10 | GCF_000235975.1 | Novosphingobium pentaromativorans US6-1 | 5 344 974 | 63.1 | 123 | 浅海沉积物 |
| 11 | GCF_000767465.1 | Novosphingobium pentaromativorans US6-1 | 5 457 578 | 63.0 | 6 | 浅海沉积物 |
| 12 | GCF_000813185.1 | Novosphingobium sp. MBES04 | 5 361 448 | 63.3 | 33 | 浅海海水 |
| 13 | GCF_001008165.2 | Croceibacterium atlanticum 26DY36 | 3 475 106 | 61.9 | 2 | 深海沉积物 |
| 14 | GCF_001010925.1 | Aurantiacibacter gangjinensis K7-2 | 2 724 959 | 62.7 | 8 | 浅海海水 |
| 15 | GCF_001010945.1 | Alteraurantiacibacter luteus KA37 | 2 887 216 | 67.2 | 22 | 浅海沉积物 |
| 16 | GCF_001013305.1 | Alteraurantiacibacter marinus HWDM-33 | 2 834 102 | 59.1 | 10 | 浅海海水 |
| 17 | GCF_001021555.1 | Alteraurantiacibacter marinus KCTC 23554 | 2 835 898 | 59.1 | 5 | 浅海海水 |
| 18 | GCF_001028625.1 | Pelagerythrobacter marensis KCTC 22370 | 2 885 033 | 64.7 | 1 | 浅海海水 |
| 19 | GCF_001077815.2 | Aurantiacibacter atlanticus s21-N3 | 3 226 359 | 58.3 | 2 | 深海沉积物 |
| 20 | GCF_001281485.1 | Altererythrobacter epoxidivorans CGMCC 1.7731 | 2 786 256 | 61.5 | 1 | 深海沉积物 |
| 21 | GCF_001461885.1 | Pelagerythrobacter marensis DSM 21428 | 2 902 055 | 64.7 | 14 | 浅海海水 |
| 22 | GCF_001634625.1 | Croceicoccus bisphenolivorans H4 | 3 602 538 | 62.8 | 74 | 浅海海水 |
| 23 | GCF_001634715.1 | Erythrobacter sp. HI0020 | 3 189 298 | 63.5 | 275 | 浅海海水 |
| 24 | GCF_001635465.1 | Erythrobacter sp. HI00D59 | 3 347 358 | 63.4 | 121 | 深海海水 |
| 25 | GCF_001635685.1 | Erythrobacter sp. HI0063 | 3 258 146 | 63.3 | 356 | 深海海水 |
| 26 | GCF_001661675.2 | Croceicoccus marinus E4A9 | 4 109 188 | 64.5 | 3 | 深海沉积物 |
| 27 | GCF_001661915.1 | Croceicoccus pelagius Ery9 | 3 306 530 | 62.8 | 40 | 浅海海水 |
| 28 | GCF_001661965.1 | Croceicoccus mobilis Ery22 | 4 210 079 | 62.5 | 32 | 深海沉积物 |
| 29 | GCF_001663155.1 | Altererythrobacter ishigakiensis NBRC 107699 | 2 673 978 | 56.9 | 1 | 浅海沉积物 |
| 30 | GCF_001677335.1 | Erythrobacter dokdonensis DSW-74 | 2 995 154 | 64.8 | 11 | 浅海海水 |
| 31 | GCF_001687545.1 | Paraurantiacibacter namhicola JCM 16345 | 2 591 679 | 65.0 | 1 | 浅海海水 |
| 32 | GCF_001698205.1 | Tsuneonella dongtanensis KCTC 22672 | 3 009 495 | 65.8 | 1 | 浅海沉积物 |
| 33 | GCF_001719165.1 | Erythrobacter litoralis DSM 8509 | 3 251 353 | 65.2 | 1 | 浅海海水 |
| 34 | GCF_001886695.1 | Aurantiacibacter gangjinensis JCM 15420 | 2 724 951 | 62.7 | 2 | 浅海海水 |
| 35 | GCF_002155305.1 | Erythrobacter dokdonensis DSM 17193 | 2 996 446 | 64.8 | 13 | 浅海海水 |
| 36 | GCF_002155425.1 | Erythrobacter donghaensis DSM 16220 | 3 372 281 | 66.2 | 11 | 浅海海水 |
| 37 | GCF_002155655.1 | Erythrobacter sanguineus JCM 20691 | 3 018 761 | 63.6 | 34 | 浅海海水 |
| 38 | GCF_002237615.1 | Qipengyuania flava VG1 | 3 184 307 | 63.3 | 1 | 浅海沉积物 |
| 39 | GCF_002269345.1 | Tsuneonella mangrovi C9-11 | 2 702 851 | 63.5 | 1 | 浅海沉积物 |
| 40 | GCF_002556635.1 | Novosphingobium sp. PC22D | 5 024 522 | 65.8 | 57 | 深海海水 |
| 41 | GCF_002795865.1 | Qipengyuania seohaensis SW-135 | 2 942 673 | 61.7 | 1 | 浅海沉积物 |
| 42 | GCF_002870965.1 | Tsuneonella flava MS1-4 | 3 275 964 | 60.5 | 29 | 浅海沉积物 |
| 43 | GCF_002895025.1 | Erythrobacter sp. SAORIC-644 | 3 549 198 | 60.5 | 25 | 深海海水 |
| 44 | GCF_003264115.1 | Erythrobacter sp. KY5 | 3 311 272 | 60.8 | 1 | 浅海海水 |
| 45 | GCF_003340475.1 | Alteripontixanthobacter maritimus HME9302 | 2 676 898 | 60.8 | 2 | 浅海海水 |
| 46 | GCF_003355455.1 | Erythrobacter aureus YH-07 | 3 372 129 | 62.0 | 2 | 浅海沉积物 |
| 47 | GCF_003363135.1 | Alteriqipengyuania lutimaris S-5 | 3 290 764 | 65.4 | 12 | 浅海海水 |
| 48 | GCF_003569745.1 | Altererythrobacter sp. B11 | 3 842 046 | 65.4 | 1 | 深海沉积物 |
| 49 | GCF_003584015.1 | Aurantiacibacter xanthus CCTCC AB 2015396 | 4 380 877 | 64.5 | 146 | 浅海海水 |
| 50 | GCF_003584125.1 | Aurantiacibacter zhengii V18 | 3 803 809 | 62.7 | 29 | 深海沉积物 |
| 51 | GCF_003605475.1 | Aurantiacibacter aquimixticola JSSK-14 | 2 552 891 | 63.0 | 2 | 浅海海水 |
| 52 | GCF_003605755.1 | Tsuneonella suprasediminis Ery12 | 3 304 416 | 60.9 | 22 | 深海海水 |
| 53 | GCF_004004515.1 | Croceicoccus ponticola GM-16 | 3 244 470 | 62.1 | 17 | 浅海海水 |
| 54 | GCF_004296255.1 | Qipengyuania flava KJ5 | 2 819 202 | 63.9 | 1 | 浅海海水 |
| 55 | GCF_004965515.1 | Alteraurantiacibacter aquimixticola SSKS-13 | 3 433 901 | 63.9 | 5 | 浅海沉积物 |
| 56 | GCF_005434975.1 | Parerythrobacter lutipelagi GH1-16 | 3 097 651 | 60.6 | 2 | 浅海沉积物 |
| 57 | GCF_005884405.1 | Qipengyuania marisflavi KEM-5 | 2 669 685 | 61.7 | 18 | 浅海海水 |
| 58 | GCF_006542605.1 | Porphyrobacter sp. YT40 | 3 943 368 | 65.8 | 2 | 浅海海水 |
| 59 | GCF_007004095.1 | Erythrobacter insulae JBTF-M21 | 2 986 343 | 57.0 | 3 | 浅海海水 |
| 60 | GCF_007570835.1 | Croceicoccus sediminis S2-4-2 | 3 548 496 | 63.0 | 9 | 浅海沉积物 |
| 61 | GCF_007830695.1 | Altererythrobacter ishigakiensis ATCC BAA-2084 | 2 672 716 | 56.9 | 4 | 浅海沉积物 |
| 62 | GCF_008107685.1 | Novosphingobium sp. BW1 | 4 460 758 | 64.5 | 134 | 浅海沉积物 |
| 63 | GCF_008932225.1 | Qipengyuania flava 21-3 | 3 229 668 | 63.6 | 1 | 深海沉积物 |
| 64 | GCF_009746585.1 | Novosphingobium aquimarinum M24A2M | 4 377 191 | 64.0 | 57 | 浅海海水 |
| 65 | GCF_009827295.1 | Qipengyuania pelagi JCM 17468 | 3 031 738 | 64.2 | 9 | 浅海海水 |
| 66 | GCF_009827315.1 | Qipengyuania gaetbuli DSM 16225 | 2 780 525 | 64.1 | 4 | 浅海沉积物 |
| 67 | GCF_009827395.1 | Pontixanthobacter aquaemixtae KCTC 52763 | 2 983 943 | 58.5 | 3 | 浅海海水 |
| 68 | GCF_009827435.1 | Croceibacterium salegens MCCC 1K01500 | 3 636 778 | 64.6 | 63 | 浅海沉积物 |
| 69 | GCF_009827445.1 | Qipengyuania aquimaris JCM 12189 | 2 662 510 | 61.8 | 1 | 浅海海水 |
| 70 | GCF_009827455.1 | Pontixanthobacter aestiaquae KCTC 42006 | 2 868 693 | 57.2 | 2 | 浅海海水 |
| 71 | GCF_009827515.1 | Pelagerythrobacter marinus H32 | 3 001 465 | 68.2 | 16 | 深海海水 |
| 72 | GCF_009827535.1 | Qipengyuania oceanensis MCCC 1A09965 | 2 874 198 | 63.9 | 14 | 深海沉积物 |
| 73 | GCF_009827545.1 | Pontixanthobacter gangjinensis JCM 17802 | 2 888 119 | 55.5 | 1 | 浅海沉积物 |
| 74 | GCF_009827615.1 | Allopontixanthobacter confluentis KCTC 52259 | 2 934 197 | 59.1 | 4 | 浅海海水 |
| 75 | GCF_009827635.1 | Parapontixanthobacter aurantiacus MCCC 1A09962 | 2 895 130 | 61.2 | 7 | 深海沉积物 |
| 76 | GCF_009828015.1 | Qipengyuania citrea CGMCC 1.8703 | 3 031 321 | 64.2 | 24 | 浅海海水 |
| 77 | GCF_009828025.1 | Qipengyuania algicida KEMB 9005-328 | 3 215 496 | 60.7 | 21 | 浅海海水 |
| 78 | GCF_009828095.1 | Pontixanthobacter luteolus SW-109 | 2 887 613 | 59.3 | 3 | 浅海海水 |
| 79 | GCF_009828115.1 | Allopontixanthobacter sediminis KCTC 42453 | 3 158 912 | 61.5 | 6 | 浅海沉积物 |
| 80 | GCF_011762005.1 | Qipengyuania flava DSM 16421 | 2 813 855 | 63.9 | 5 | 浅海海水 |
| 81 | GCF_012972675.1 | Pontixanthobacter rizhaonensis RZ02 | 2 790 898 | 55.5 | 4 | 浅海海水 |
| 82 | GCF_012979275.1 | Pseudopontixanthobacter vadosimaris JL3514 | 2 629 682 | 63.0 | 43 | 浅海海水 |
| 83 | GCF_013260645.1 | Erythrobacter mangrovi EB310 | 3 058 693 | 62.9 | 1 | 浅海沉积物 |
| 84 | GCF_013370205.1 | Actirhodobacter atriluteus HHU K3-1 | 2 939 611 | 62.1 | 23 | 浅海海水 |
| 85 | GCF_013371495.1 | Altererythrobacter lutimaris JGD-16 | 2 952 860 | 57.8 | 33 | 浅海海水 |
| 86 | GCF_013408095.1 | Novosphingobium marinum DSM 29043 | 3 591 024 | 64.2 | 22 | 深海海水 |
| 87 | GCF_014191645.1 | Alteriqipengyuania lutimaris CECT 8624 | 3 269 205 | 65.5 | 6 | 浅海沉积物 |
| 88 | GCF_014199315.1 | Croceibacterium atlanticum DSM 100738 | 3 457 495 | 61.9 | 22 | 深海沉积物 |
| 89 | GCF_014640055.1 | Novosphingobium marinum CGMCC 1.12918 | 3 590 209 | 64.2 | 27 | 深海海水 |
| 90 | GCF_014641655.1 | Alteriqipengyuania_A marina CGMCC 1.15297 | 2 634 902 | 64.9 | 56 | 浅海海水 |
| 91 | GCF_014642495.1 | Croceicoccus pelagius CGMCC 1.15358 | 3 228 892 | 62.8 | 42 | 浅海海水 |
| 92 | GCF_014642595.1 | Croceicoccus mobilis CGMCC 1.15360 | 4 116 752 | 62.6 | 37 | 深海沉积物 |
| 93 | GCF_014645195.1 | Novosphingobium indicum CGMCC 1.6784 | 4 961 652 | 63.0 | 103 | 深海沉积物 |
| 94 | GCF_014705715.1 | Erythrobacter rubeus KMU-140 | 3 042 712 | 60.6 | 4 | 浅海海水 |
| 95 | GCF_015529805.1 | Qipengyuania soli 6D36 | 2 910 144 | 63.3 | 1 | 浅海沉积物 |
| 96 | GCF_015865035.1 | Novosphingobium aureum YJ-S2-02 | 4 342 390 | 65.5 | 39 | 浅海沉积物 |
| 97 | GCF_015999305.1 | Erythrobacter sp. YJ-T3-07 | 3 317 720 | 64.3 | 494 | 浅海沉积物 |
| 98 | GCF_016019885.1 | Aurantiacibacter sediminis JGD-13 | 2 668 619 | 60.2 | 14 | 浅海沉积物 |
| 99 | GCF_016745095.1 | Croceicoccus sp. YJ47 | 3 353 209 | 64.4 | 1 | 深海海水 |
| 100 | GCF_017312445.1 | Novosphingobium sp. PY1 | 4 764 096 | 63.4 | 36 | 浅海沉积物 |
| 101 | GCF_018205975.1 | Erythrobacter sp. JK5 | 3 340 457 | 64.2 | 1 | 浅海沉积物 |
| 102 | GCF_018398395.1 | Croceicoccus gelatinilyticus 1NDH52 | 3 790 455 | 62.6 | 43 | 浅海沉积物 |
| 103 | GCF_018417475.1 | Novosphingobium decolorationis 502str22 | 4 750 375 | 65.5 | 2 | 深海沉积物 |
| 104 | GCF_018636735.1 | Erythrobacter sp. 3-20A1M | 3 116 220 | 64.8 | 1 | 浅海海水 |
| 105 | GCF_900115585.1 | Qipengyuania nanhaisediminis CGMCC 1.7715 | 2 899 438 | 62.0 | 12 | 浅海沉积物 |
| 106 | GCF_900143235.1 | Erythrobacter sanguineus DSM 11032 | 2 978 798 | 63.8 | 25 | 浅海海水 |
| 107 | GCF_900177715.1 | Altererythrobacter xiamenensis CGMCC 1.12494 | 3 082 408 | 61.8 | 5 | 浅海海水 |